FateCompass : identifier et prédire les critères décisifs du destin cellulaire
La différenciation cellulaire est un processus régulé par l’expression des gènes grâce à l’action de facteurs de transcription. Différents facteurs de transcription et des changements dans l’ordre d’activation des gènes mènent à des destins cellulaires variés. Dans une étude publiée dans Cell Reports Methods, les équipes de Nacho Molina et de Gérard Gradwohl ont développé FateCompass, un outil capable d’identifier et prédire l’impact de ces différences dans la spécification des cellules.
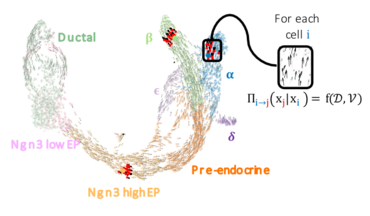
Au cours du processus de différenciation cellulaire, les gènes sont activés et désactivés à des moments précis par des facteurs de transcription, ce qui permet d’obtenir une diversité cellulaire. Comprendre les critères déterminants du destin cellulaire permet d’apporter un éclairage dans de nombreux domaines comme l’embryogénèse, la médecine régénérative, les thérapies géniques et cellulaires, les cancers… Actuellement, pour déterminer les facteurs impactant le destin cellulaire, on compare les niveaux d’expression des facteurs de transcription et de leurs gènes cibles. Cependant, il est difficile de mesurer avec précision l'activité régulatrice dynamique des facteurs de transcription au niveau des cellules individuelles et à haut débit.
Dans cette étude, les scientifiques présentent leur outil FateCompass, une approche qui exploite les données transcriptomiques d'une cellule pour identifier les facteurs de transcription spécifiques à des lignages cellulaires au cours du processus de différenciation. Pour le développer, les chercheurs se sont appuyés sur des jeux de données transcriptomiques issues de cellules d’îlots de Langerhans en formation au cours du développement pancréatique chez la souris, ou dérivées, in vitro, de cellules souches humaines. L’outil estime la probabilité de transition entre différents états cellulaires, en combinant une approche probabiliste avec la vitesse de maturation des ARNm ou le potentiel de différenciation. En considérant les changements dynamiques et les corrélations entre l'expression des gènes et les activités des facteurs de transcription, FateCompass identifie efficacement les régulateurs qui sont spécifiques à des lignages particuliers. Ainsi, l’outil a pu correctement identifier des régulateurs spécifiques déjà connus de la formation des cellules ß sécrétrices d’insuline et a révélé l’existence de nouveaux régulateurs potentiels.
Avec FateCompass, cette étude met à disposition un nouvel outil précieux pour générer des hypothèses et améliorer la compréhension des réseaux de régulation génique qui déterminent le destin cellulaire.

FateCompass est un nouvel outil qui aide les chercheurs à identifier les régulateurs spécifiques impliqués dans les processus de différenciation cellulaire. En analysant les données transcriptomiques d'une seule cellule, il estime l'activité dynamique des facteurs de transcription et prédit les régulateurs spécifiques à un lignage, améliorant ainsi notre compréhension des réseaux de régulation génique qui régissent les décisions relatives au destin des cellules.
Crédits : Sacha Jimenez, IGBMC
