
GenomEast
GenomEast
RESPONSABLE DE PLATEFORME
Forts d'une expérience de plus de 20 ans, nous proposons une large gamme de services pour explorer les génomes, leur expression et leur régulation, du contrôle qualité des échantillons de départ jusqu'à l'analyse des données. Ces services sont destinés à l'ensemble de la communauté scientifique, nationale et internationale, publique et privée.




La plateforme est membre du noyau central de l'Infrastructure Nationale de Recherche France Génomique.
La plateforme est labellisée IBISA.
La plateforme est certifiée ISO 9001 - NF X 50-900.
Nos prestations
Nos prestations et nos recommandations sont détaillées dans les fiches produits ci-contre.
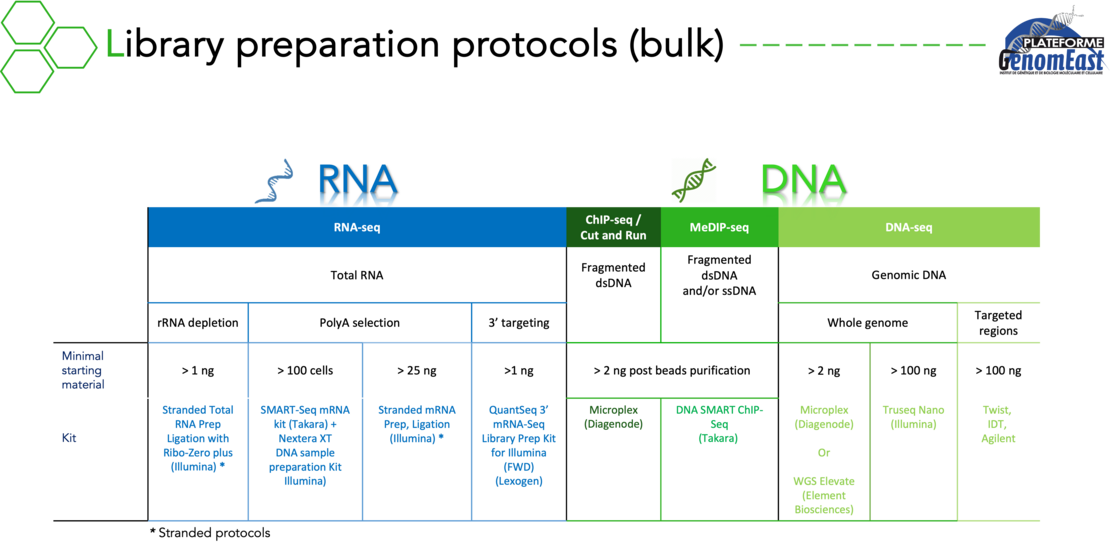
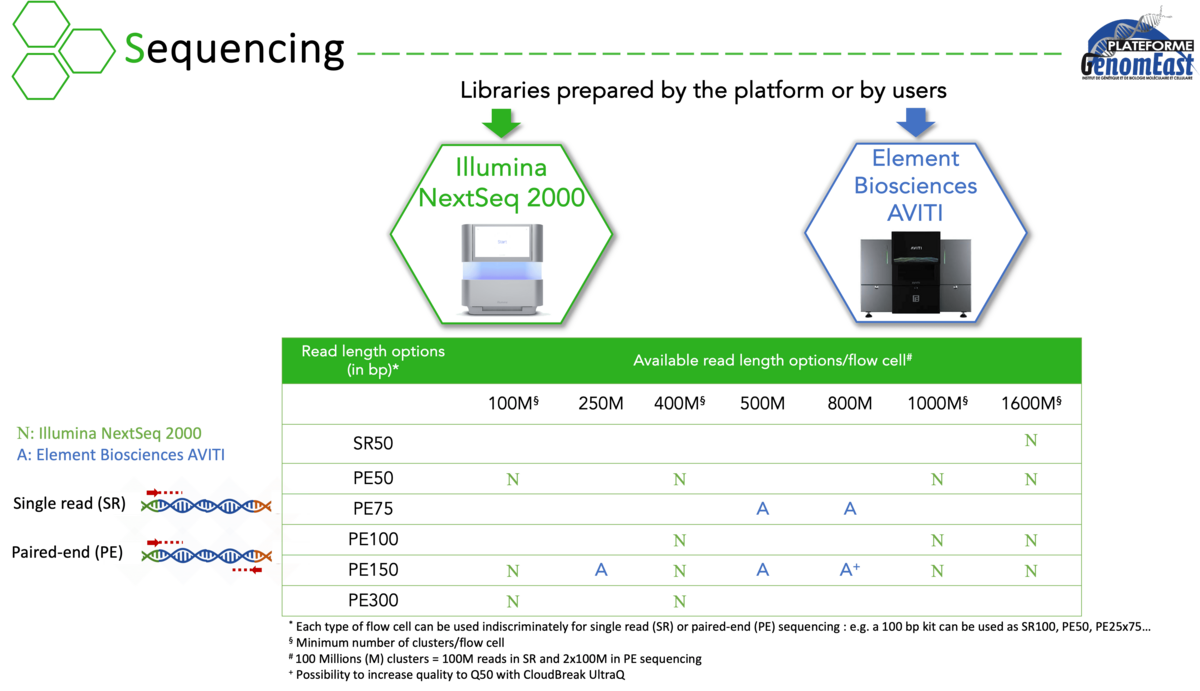
Actuellement équipés des séquenceurs Illumina NextSeq 2000 et Element Biosciences AVITI, nous réalisons des projets d'analyse :
du transcriptome :
- RNA-seq
- Single cell RNA-seq (scRNA-seq)
- Transcriptomique spatiale
de l'épigénome :
- ChIP-seq
- Cut & Run
- MeDIP-seq
- Nous avons également de l'expérience dans le séquençage de vos librairies ATAC-seq ou Cut & Tag
multiomique :
- Expression des gènes + ATAC en cellule unique
Pour ces projets, nous préparons et séquençons vos librairies en réalisant des contrôles qualité à chaque étape. Nous pouvons également réaliser l’analyse bioinformatique de vos données. Nous avons une expertise dans une grande variété d’applications et nous avons développé des pipelines bioinformatiques dédiés pour analyser les résultats correspondants.
Alternativement, vous pouvez utiliser notre capacité de séquençage pour séquencer vos propres libraires dans la mesure où elles sont compatibles avec la technologie Illumina.
Fiches produits
- Transcriptomique
- Epigénomique
- Multiomique
- Séquençage de vos librairies
Besoin d'aide pour le design de votre expérience ?
Consultez nos fiches produit ci-dessus ou nos schémas d’aide à la décision ci-après :
Librairies
Cellules / Noyaux uniques

Transcriptomique spatiale

Séquençage
Des questions ?
N’hésitez pas à nous contacter (prenom.nom@igbmc.fr) :
Pour les projets portant sur l’ARN : Christelle Thibault et Céline Keime
Pour les projets portant sur l’ADN : Bernard Jost et Tao Ye
Soumettre un projet
Comment soumettre un projet à GenomEast ?
Via notre interface web
Quelle procédure suivre ?
Comment envoyer des échantillons à GenomEast ?
Tous nos services sont régis par les conditions générales de vente qui prévalent sur toutes les conditions d'achat, sauf accord écrit de notre part. Vous pouvez également consulter notre plan de gestion des données.
Formations
Nous partageons régulièrement nos connaissances et expertises à travers différents enseignements et formations, notamment :
L’UE « Omique 1 » du Master 1 Biologie-Santé, parcours Recherche en Biomédecine à l’Université de Strasbourg
l'UE « Omiques avancées » du Master 2 Bioinformatique et bioimagerie structurale à l'Université de Strasbourg
L'EC « Méthodologie R&D : de l'atome aux modèles précliniques » du Master 2 Ingénierie Biologique et Applications thérapeutiques à l'Université de Reims
Le PhD Program de l’école Universitaire de Recherche IMCBio à Strasbourg
L'école de bioinformatique AVIESAN IFB Inserm
L’organisation de formations en bioinformatique pour le séquençage haut débit avec l’Inserm et le CNRS
Membres
Responsable de la plateforme
Christelle Thibault-Carpentier
Traitement des échantillons
- Bernard Jost (responsable)
- Marie Cerciat
- Amal El Amri
- Romain Kaiser
- Christelle Morgenthaler-Roth
- Angélique Pichot
- David Rodriguez
- Serge Vicaire
Bioinformatique
- Céline Keime (responsable)
- Matthieu Jung
- Damien Plassard
- Tao Ye
Ressources
La plateforme est équipée de technologies de pointe parmi lesquelles :

Le séquenceur haut débit NextSeq 2000 (Illumina)

Le séquenceur haut débit AVITI (Element Biosciences)

La technologie en cellule unique du Chromium iX (10x Genomics)

La technologie spatial Visium CytAssist (10X Genomics)
Nos autres équipements:
- Séquençage haut débit : iSeq100 (Illumina)
- Transcriptomique spatiale : GeoMx Digital Spatial Profiler (NanoString)
- Contrôle qualité : Flash Reader Varioskan et Qubit Fluorometer (Thermo Fisher), 2100 Bioanalyzer et Fragment Analyzer AATI (Agilent)
- Sonicateur : E220 AFA system (Covaris)
- Infrastructure informatique : calcul (Dell) : 592 cœurs, stockage (Lenovo, GPFS) : 750 To
Réseaux et financements
France Génomique
Notre plateforme est partenaire du réseau France Génomique depuis la sélection initiale du projet dans le cadre des « Investissements d'avenir » en 2011. Ce réseau rassemble et partage les ressources des principales plateformes françaises de génomique et de bioinformatique.
Fondation maladies rares
Depuis 2011, nous sommes l’une des plateformes partenaires de la Fondation Maladies Rares pour la réalisation du programme « GenOmics : séquençage à haut débit & maladies rares » visant à élucider les bases génétiques et moléculaires des maladies rares en s’appuyant sur le séquençage à haut débit.
Notre plateforme a bénéficié d’un soutien financier de nombreux partenaires pour l’acquisition d’équipements et de technologies nécessaires à son développement et pour le financement de personnels, que nous remercions.

Nouvelles applications et technologies
Au cours des dernières années, nous avons acquis une solide expérience dans les technologies de séquençage pour cellule unique (prix « Expertises Recherche » 2021 de l’Université de Strasbourg). Nous continuons à implémenter de nouvelles applications chaque année. En parallèle, nous travaillons à la mise en œuvre de deux technologies de transcriptomique spatiale complémentaires, les technologies Visium de 10X Genomics et GeoMx de NanoString.
Publications
2025
Article dans une revue
Upregulation of adenosine A<sub>2A</sub> receptor in astrocytes is sufficient to trigger hippocampal multicellular dysfunctions and memory deficits
- Agathe Launay
- Kevin Carvalho
- Athénais Genin
- Thibaut Gauvrit
- Paola Nobili
- Victoria Gomez-Murcia
- Emma Augustin
- Anaëlle Burgard
- Johanne Gambi
- Déborah Fourmy
- Bryan Thiroux
- Didier Vieau
- Alexis-Pierre Bemelmans
- Stephanie Le Gras
- Luc Buée
- Miranda E Orr
- Etienne Audinat
- Anne-Laurence Boutillier
- Gilles Bonvento
- Karine Cambon
- ...
Molecular Psychiatry ; Volume: 30 ; Page: 5300-5314
Article dans une revue
Compromised retinoic acid receptor beta expression accelerates the onset of motor, cellular and molecular abnormalities in a mouse model of Huntington’s disease
- Nicolas Zinter
- Tao Ye
- Hanna Semaan
- Valérie Fraulob
- Damien Plassard
- Wojciech Krezel
Neurobiology of Disease ; Volume: 212 ; Page: 106943
Assessment of the effects of estetrol and 17α-ethinylestradiol on zebrafish (Danio rerio) metamorphosis: a morphological and transcriptomic approach
- Nathalie Leroux
- Sébastien Baekelandt
- Jean-Baptiste Robert
- Laura Burattin
- Céline Keime
- Céline Gérard
- Patrick Kestemont
Elsevier BV ; Volume: 376 ; Page: 126423
The transcription factor IID subunit Taf13 is dispensable for TATA binding protein promoter recruitment and RNA polymerase II transcription
- Igor Martianov
- Stephanie Le Gras
- Guillaume Davidson
- Irwin Davidson
Elsevier BV ; Volume: 28 ; Page: 112286
Article dans une revue
Hippocampal Astrocyte Morphology Follows an Unexpected Trajectory With Age in a Transgenic Rodent Model of Tauopathy.
- Emma Augustin
- Tatiana Vinasco-Sandoval
- Miriam Riquelme-Perez
- Damien Plassard
- Mylène Gaudin
- Gwenaëlle Aurégan
- Julien Mitja
- Sueva Bernier
- Charlène Joséphine
- Fanny Petit
- Caroline Jan
- Anne Sophie Hérard
- Marie-Claude Gaillard
- Agathe Launay
- Emilie Faivre
- Luc Buee
- Anne Laurence Boutillier
- David Blum
- Alexis-Pierre Bemelmans
- Gilles Bonvento
- ...
Glia ; Volume: 73 ; Page: 1502-1519
TFE3 fusions drive oxidative metabolism and ferroptosis resistance in translocation renal cell carcinoma
- Alexandra Helleux
- Guillaume Davidson
- Antonin Lallement
- Fatima Al Hourani
- Alexandre Haller
- Isabelle Michel
- Anas Fadloun
- Christelle Thibault-Carpentier
- Xiaoping Su
- Véronique Lindner
- Thibault Tricard
- Hervé Lang
- Nizar M Tannir
- Irwin Davidson
- Gabriel G Malouf
Springer Science and Business Media LLC ; Volume: 17 ; Page: 1041-1070
CREB3 gain of function variants protect against ALS
- Salim Megat
- Christine Marques
- Marina Hernán-Godoy
- Chantal Sellier
- Geoffrey Stuart-Lopez
- Sylvie Dirrig-Grosch
- Charlotte Gorin
- Aurore Brunet
- Mathieu Fischer
- Céline Keime
- Pascal Kessler
- Marco Antonio Mendoza-Parra
- Ramona A. J. Zwamborn
- Jan H. Veldink
- Sonja W. Scholz
- Luigi Ferrucci
- Albert Ludolph
- Bryan Traynor
- Adriano Chio
- Luc Dupuis
- ...
Springer Science and Business Media LLC ; Volume: 16
Article dans une revue
Negative energy balance by feed deprivation affects the adipose miRNome in the lactating goat
- Yannick Faulconnier
- Tao Ye
- Christine Leroux
Journal of Animal Science ; Volume: 103 ; Page: skaf044
Article dans une revue
Gene-specific transcript buffering revealed by perturbation of coactivator complexes
- Faezeh Forouzanfar
- David F Moreno
- Damien Plassard
- Audrey Furst
- Karen Amaral de Oliveira
- Bernardo Reina San Martin
- Làszlo Tora
- Nacho Molina
- Manuel Mendoza
Science Advances ; Volume: 11 ; Page: eadr1492
Article dans une revue
Transcriptomic analysis of human primary T cells after short-term leucine-deprivation and evaluation of kinase GCN2’s role in regulating differential gene expression
- Aurore Dougé
- Gwendal Cueff
- Céline Keime
- Valérie Carraro
- Céline Jousse
- Paul Rouzaire
- Alain Bruhat
PLoS ONE ; Volume: 20 ; Page: e0317505
Page 1 sur 10

