Maturation des ARN messagers
Maturation des ARN messagers
L’ADN contient l’information nécessaire à la synthèse des protéines, qui sont les principaux éléments architecturaux et effecteurs des organismes vivants. Cette information n'est toutefois pas directement déchiffrée à partir de l'ADN par la machinerie de synthèse des protéines. Elle est tout d'abord extraite sous forme de copies transitoires et mobiles du matériel génétique : les ARN messagers (ARNm).
L'utilisation d'une molécule intermédiaire comme vecteur protège non seulement la précieuse information héréditaire de l'ADN, mais permet également le contrôle precis de l'expression des gènes.
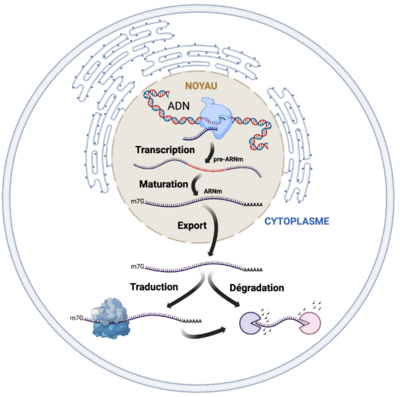
Le cycle de vie des ARNm eucaryotes est complexe. Ils sont synthétisés lors de la transcription par l'ARN polymérase II et modifiés co/post-transcriptionnellement pendant l’étape de maturation. Les ARNm « matures » sont ensuite exportés dans le cytoplasme pour être décodés en protéines par le ribosome, lors de la traduction. Enfin, les transcrits sont éliminés par des voies de dégradation spécifiques de l'ARNm.
L’équipe s’intéresse à l’étape de maturation des ARNm. En particulier, elle cherche à déterminer les mécanismes moléculaires des machines moléculaires qui modifient les ARNm, changeant leur stabilité, leur « traductibilité » ou encore l’information génétique qu’ils contiennent !
Pour cela, les complexes moléculaires d’intérêt, composés de protéines et/ou d’ARN sont reconstitués à partir de sources recombinantes et/ou d’extrait cellulaire. Leurs compositions et structures chimiques sont ensuite étudiées par des approches biochimiques, biophysiques et de biologie structurale (Cryo-microscopie électronique (cryo-EM), en particulier).
Projets en cours
Fidélité de l'épissage
Lors de l’épissage, le spliceosome identifie puis élimine certaines séquences présentes au sein des précurseurs d’ARN messagers (pre-ARNm), les introns. Pour un pre-ARNm donné, les séquences éliminées peuvent varier en fonction du type cellulaire ou de divers signaux. Ce phénomène « d’épissage alternatif » permet, à partir d’un seul et même gène, de produire plusieurs types de protéines ! L'épissage doit être réalisé sans erreur pour éviter la production d’ARNm aberrants, codant des protéines potentiellement toxiques. La fidélité de l'épissage repose sur la précision du spliceosome, une machinerie moléculaire de grande taille composée de dizaines de protéines et d’ARN, qui définit les limites des introns avant de catalyser leurs excisions.
Ce projet vise à déterminer comment le spliceosome sélectionne les introns sans commettre d’erreur, tout en montrant une grande souplesse pour permettre les changements d’épissage lors de l’épissage alternatif. Pour cela, le spliceosome humain sera isolé aux moments clés où il reconnait ses séquences cibles afin d’étudier sa composition et sa structure tridimensionnelle.
Plusieurs postes (thèse, postdoc) seront bientôt à pourvoir dans le cadre de ce projet. Les biochimistes/biologistes structuraux intéressés par la structure et la fonction des grand assemblages protéines/ARN peuvent contacter Clément Charenton directement.
Financements et partenaires
ERC Starting Grant

ATIP-avenir

Actualités

Une nouvelle équipe de recherche spécialisée dans la maturation de l'ARN
Clément Charenton, accueilli temporairement dans l’équipe d’Albert Weixlbaumer, démarre sa propre équipe de recherche à l’IGBMC au sein du département…
Lire la suite
Membres
Chercheur(euse)s
Doctorant(e)s
Ingénieur(eure)s
Publications
2025
Pré-publication, Document de travail » Preprint/Prepublication
FAIMS-GPF XL-MS: crosslinking-mass spectrometry based on gas-phase fractionation
- Hugo Gizardin-Fredon
- Salvatore Terrosu
- Simon Pichard
- Dursun Korkut
- Cathy Braun
- Arnaud Poterszman
- Clément Charenton
- Sarah Cianférani
Article dans une revue
Dominant variants in major spliceosome U4 and U5 small nuclear RNA genes cause neurodevelopmental disorders through splicing disruption
- Caroline Nava
- Benjamin Cogne
- Amandine Santini
- Elsa Leitão
- François Lecoquierre
- Yuyang Chen
- Sarah Stenton
- Thomas Besnard
- Solveig Heide
- Sarah Baer
- Abhilasha Jakhar
- Sonja Neuser
- Boris Keren
- Anne Faudet
- Sylvie Forlani
- Marie Faoucher
- Kevin Uguen
- Konrad Platzer
- Alexandra Afenjar
- Jean-Luc Alessandri
- ...
Nature Genetics
2020
Article dans une revue
Pby1 is a direct partner of the Dcp2 decapping enzyme
- Clément Charenton
- Claudine Gaudon-Plesse
- Régis Back
- Nathalie Ulryck
- Loreline Cosson
- Bertrand Séraphin
- Marc Graille
Nucleic Acids Research ; Volume: 48
2017
Article dans une revue
A unique surface on Pat1 C-terminal domain directly interacts with Dcp2 decapping enzyme and Xrn1 5′–3′ mRNA exonuclease in yeast
- Clément Charenton
- Claudine Gaudon-Plesse
- Zaineb Fourati
- Valério Taverniti
- Régis Back
- Olga Kolesnikova
- Bertrand Séraphin
- Marc Graille
Proceedings of the National Academy of Sciences of the United States of America ; Volume: 114 ; Page: E9493-E9501
2016
Article dans une revue
Structure of the active form of Dcp1–Dcp2 decapping enzyme bound to m7GDP and its Edc3 activator
- Clément Charenton
- Valerio Taverniti
- Claudine Gaudon-Plesse
- Régis Back
- Bertrand Séraphin
- Marc Graille
Nature Structural and Molecular Biology ; Volume: 23 ; Page: 982-986
2014
Article dans une revue
The C-Terminal Domain from S. cerevisiae Pat1 Displays Two Conserved Regions Involved in Decapping Factor Recruitment
- Zaineb Fourati
- Olga A Kolesnikova
- Régis Back
- Jenny Keller
- Clément Charenton
- Valério Taverniti
- Claudine Gaudon Plesse
- Noureddine Lazar
- Dominique Durand
- Herman van Tilbeurgh
- Bertrand Séraphin
- Marc Graille
PLoS ONE ; Volume: 9 ; Page: e96828
