Organisation spatiale du génome
Organisation spatiale du génome
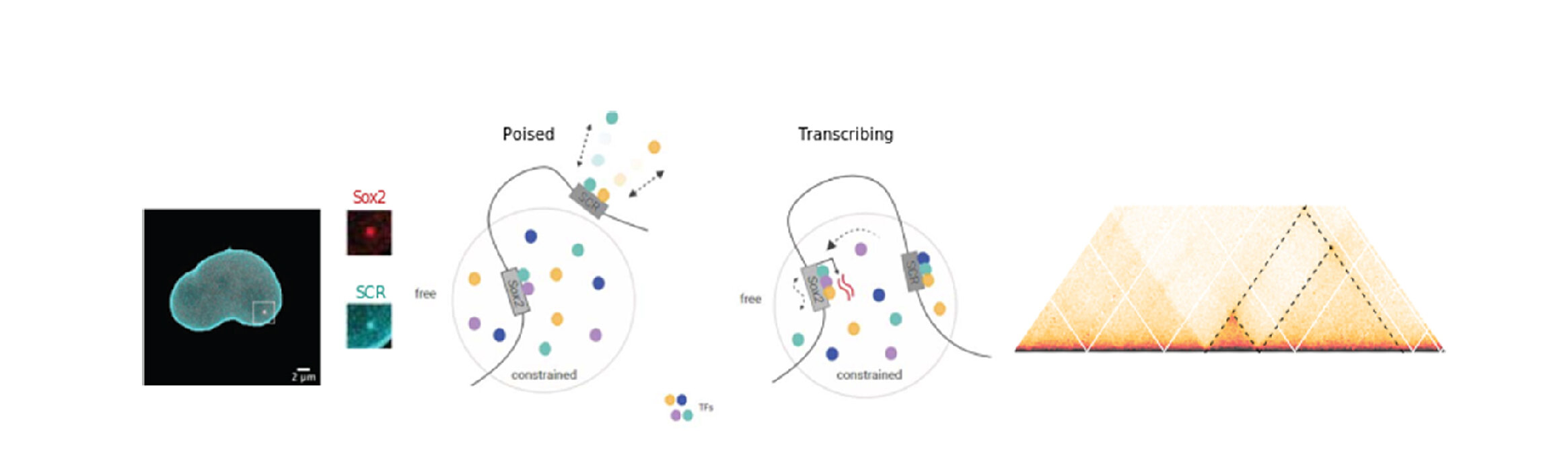
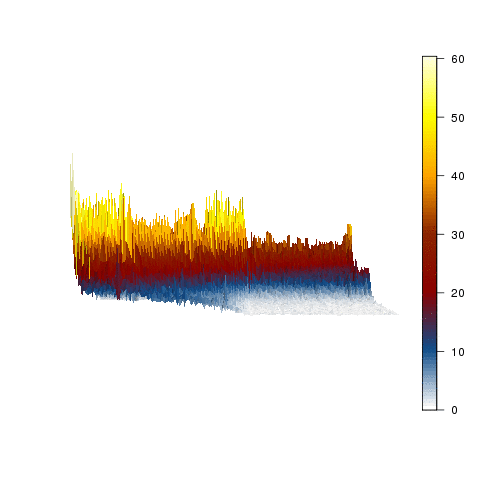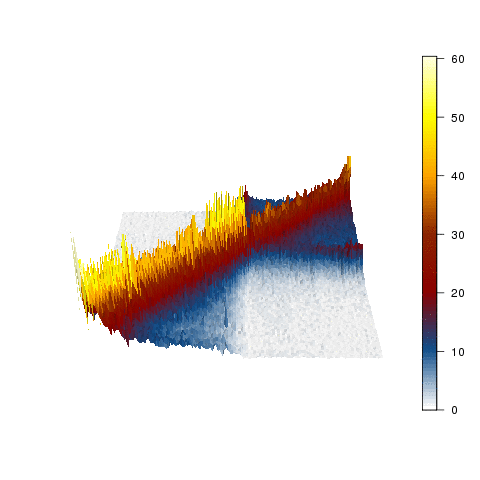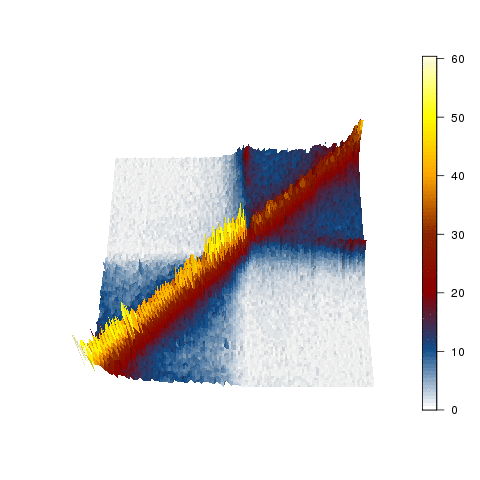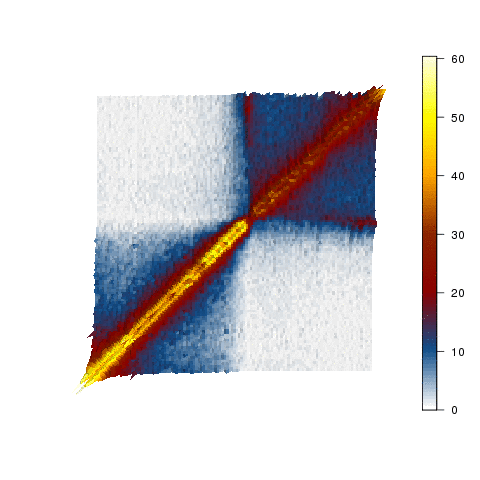
Chaque cellule du corps humain doit compacter deux mètres d'ADN dans un noyau d'environ dix micromètres, ce qui équivaut à faire tenir la Tour Eiffel dans une pièce de un centime. D'une manière ou d'une autre, ceci est réalisé de façon à ce que les gènes nécessaires à chaque cellule soient accessibles à la machinerie pour leur activation. Notre équipe combine des approches génomiques (tels qu Hi-C et ses variantes), l'édition du génome par CRISPR-Cas9 et la microscopie à haute résolution temporelle pour disséquer l'organisation du génome dans l'espace et le temps, et pour voir comment elle est responsable du contrôle de l'expression des gènes.
Twitter : @TomSexton_lab
Membres
Chercheur(euse)s
Doctorant(e)s
Ingénieur(eure)s
Collaborateur(trice)s occasionnel(le)s
Anciens membres
Yousra Ben Zouari (PhD) – en postdoc au FMI, Bâle.
Nezih Karasu (PhD) – en postdoc à l’IGMM, Montpellier
Angeliki Platania (PhD)
Sanjay Chahar (postdoc) – en postdoc a l’IGMM, Montpellier
Anne Molitor (postdoc) – poste d’Ingénieur de Recherche à l’Institut d’Immuno-Rhumathologie Moléculaire, Strasbourg
Natalia Sikorska (Postdoc) – suit une formation médicale
Gui Oliveira (Postdoc) – a accédé à un poste d'ingénieur logiciel chez MathWorks
Dominique Kobi (ingénieur) – a accédé à un poste chez Machery-Nagel
Manon Maroquenne (ingénieur) – a accédé à un poste au laboratoire de Bioingénerie et Bio-imagerie des tissus ostéo-articulaires, Paris
Audrey Mossler (ingénieur) – est au Imperial College, UK
Projets en cours
Nous nous intéressons à tous les aspects de l'organisation 3D ou 4D du génome qui influencent l'expression des gènes, mais nos recherches actuelles peuvent être divisées en deux axes principaux :
- Des approches de biologie moléculaire à l'échelle du génome (par exemple Capture Hi-C) pour caractériser les changements épigénomiques accompagnant les transitions développementales et les réponses aux traitements chimiothérapeutiques.
- L’étiquetage de loci génomiques spécifiques et leur suivi en temps réel pour voir comment la mobilité de la chromatine affecte (et est affectée par) la transcription et les réponses de réparation de l'ADN.
Notre première tentative de modélisation du noyau des cellules souches.
Collaborations et réseaux
A l’IGBMC
- Nacho Molina
- Daniel Metzger
A Strasbourg
- Karine Merienne (Laboratoire de Neurosciences Cognitives et Adaptives)
- Jean-Noel Freund/Claire Domon-Dell (Interface de Recherche Fondamentale et Appliquée Research en Cancerologie)
En France
- Kerstin Bystricky (Centre de Biologie Intégrative, Toulouse)
- Christophe Ginestier/Emmanuelle Charafe-Jauffret (Centre de Cancérologie de Marseille, Marseille)
- Mounia Lagha (Institut de génétique moléculaire, Montpellier)
- Reini Luco (Institut Curie, Paris)
- Salvatore Spicuglia (Théories and Approches de la complexité génomique, Marseille)
- Jean-Christophe Andrau (Institut de génétique moléculaire, Montpellier)
- Daniel Jost (Ecole Normale Superieur, Lyon)
A l’international
- Jennifer Mitchell (University of Toronto, Canada)
- Tineke Lenstra (National Cancer Institute, Netherlands)
- Akis Papantonis (University of Goettingen, Germany)
- Evi Soutoglou (Genome Damage and Stability Centre, UK)
Financements et partenaires
ERC Starting Grant 2015-2021;
ATIP-Avenir 2014-2017;
ANR; INCa; USIAS
Prix/Distinctions
Prix Claude Paoletti, 2015
Ressources
Voir nos outils Github
Pour l’analyse et l’exploration de Capture Hi-C (ChiCMaxima)
Pour l’exploration des données de 4C (4See)
Pour la mesure des propriétés de diffusion à partir des résultats d’imagerie en temps réél (GPTool)
Publications
2024
Article dans une revue
Gene-to-gene coordinated regulation of transcription and alternative splicing by 3D chromatin remodeling upon NF-κB activation
- Paul Marie
- Matéo Bazire
- Julien Ladet
- Lamya Ben Ameur
- Sanjay Chahar
- Nicolas Fontrodona
- Tom Sexton
- Didier Auboeuf
- Cyril F Bourgeois
- Franck Mortreux
Nucleic Acids Research ; Volume: 52 ; Page: 1527-1543
Article dans une revue
Transcription processes compete with loop extrusion to homogenize promoter and enhancer dynamics
- Angeliki Platania
- Cathie Erb
- Mariano Barbieri
- Bastien Molcrette
- Erwan Grandgirard
- Marit de Kort
- Wim Pomp
- Karen Meaburn
- Tiegh Taylor
- Virlana Shchuka
- Silvia Kocanova
- Mariia Nazarova
- Guilherme Monteiro Oliveira
- Jennifer Mitchell
- Evi Soutoglou
- Tineke Lenstra
- Nacho Molina
- Argyris Papantonis
- Kerstin Bystricky
- Tom Sexton
Science Advances ; Volume: 10 ; Page: eadq0987
2023
Article dans une revue
Transcription induces context-dependent remodeling of chromatin architecture during differentiation
- Sanjay Chahar
- Yousra Ben Zouari
- Hossein Salari
- Dominique Kobi
- Manon Maroquenne
- Cathie Erb
- Anne Molitor
- Audrey Mossler
- Nezih Karasu
- Daniel Jost
- Tom Sexton
PLoS Biology ; Volume: 21 ; Page: e3002424
Article dans une revue
Short tandem repeats are important contributors to silencer elements in T cells
- Saadat Hussain
- Nori Sadouni
- Dominic van Essen
- Lan T M Dao
- Quentin Ferré
- Guillaume Charbonnier
- Magali Torres
- Frederic Gallardo
- Charles-Henri Lecellier
- Tom Sexton
- Simona Saccani
- Salvatore Spicuglia
Nucleic Acids Research ; Volume: 51 ; Page: 4845-4866
Pré-publication, Document de travail
Competition between transcription and loop extrusion modulates promoter and enhancer dynamics
- Angeliki Platania
- Cathie Erb
- Mariano Barbieri
- Bastien Molcrette
- Erwan Grandgirard
- Marit Ac de Kort
- Karen Meaburn
- Tiegh Taylor
- Virlana M Shchuka
- Silvia Kocanova
- Guilherme Monteiro Oliveira
- Jennifer A Mitchell
- Evi Soutoglou
- Tineke L Lenstra
- Nacho Molina
- Argyris Papantonis
- Kerstin Bystricky
- Tom Sexton
- Guilherme Monteiro Oliveira
2022
Pré-publication, Document de travail
Context-dependent transcriptional remodeling of TADs during differentiation
- Sanjay Chahar
- Yousra Ben Zouari
- Hossein Salari
- Anne Molitor
- Dominique Kobi
- Manon Maroquenne
- Cathie Erb
- Audrey Mossler
- Nezih Karasu
- Daniel Jost
- Tom Sexton
Article dans une revue
Concurrent CDX2 cis -deregulation and UBTF::ATXN7L3 fusion define a novel high-risk subtype of B-cell ALL
- Marie Passet
- Rathana Kim
- Stéphanie Gachet
- François Sigaux
- Julie Chaumeil
- Ava Galland
- Thomas Sexton
- Samuel Quentin
- Lucie Hernandez
- Lise Larcher
- Hugo Bergugnat
- Tao Ye
- Nezih Karasu
- Aurélie Caye
- Beate Heizmann
- Isabelle Duluc
- Patrice Chevallier
- Philippe Rousselot
- Françoise Huguet
- Thibaut Leguay
- ...
Blood ; Volume: 139 ; Page: 3505-3518
Article dans une revue
The histone variant macroH2A1.1 regulates RNA polymerase II-paused genes within defined chromatin interaction landscapes
- Ludmila Recoules
- Alexandre Heurteau
- Flavien Raynal
- Nezih Karasu
- Fatima Moutahir
- Fabienne Bejjani
- Isabelle Jariel-Encontre
- Olivier Cuvier
- Thomas Sexton
- Anne-Claire Lavigne
- Kerstin Bystricky
Journal of Cell Science ; Volume: 135 ; Page: jcs259456
2021
Article dans une revue
Age-related and disease locus-specific mechanisms contribute to early remodelling of chromatin structure in Huntington’s disease mice
- Rafael Alcalá-Vida
- Jonathan Seguin
- Caroline Lotz
- Anne Molitor
- Ibai Irastorza-Azcarate
- Ali Awada
- Nezih Karasu
- Aurélie Bombardier
- Brigitte Cosquer
- Jose Luis Gomez Skarmeta
- Jean-Christophe Cassel
- Anne-Laurence Boutillier
- Thomas Sexton
- Karine Merienne
Nature Communications ; Volume: 12 ; Page: 364
Article dans une revue
Precise measurements of chromatin diffusion dynamics by modeling using Gaussian processes
- Guilherme Oliveira
- Attila Oravecz
- Dominique Kobi
- Manon Maroquenne
- Kerstin Bystricky
- Thomas Sexton
- Nacho Molina
Nature Communications ; Volume: 12 ; Page: 6184
Page 1 sur 3
