Régulation génomique et épigénomique du destin cellulaire
Régulation génomique et épigénomique du destin cellulaire
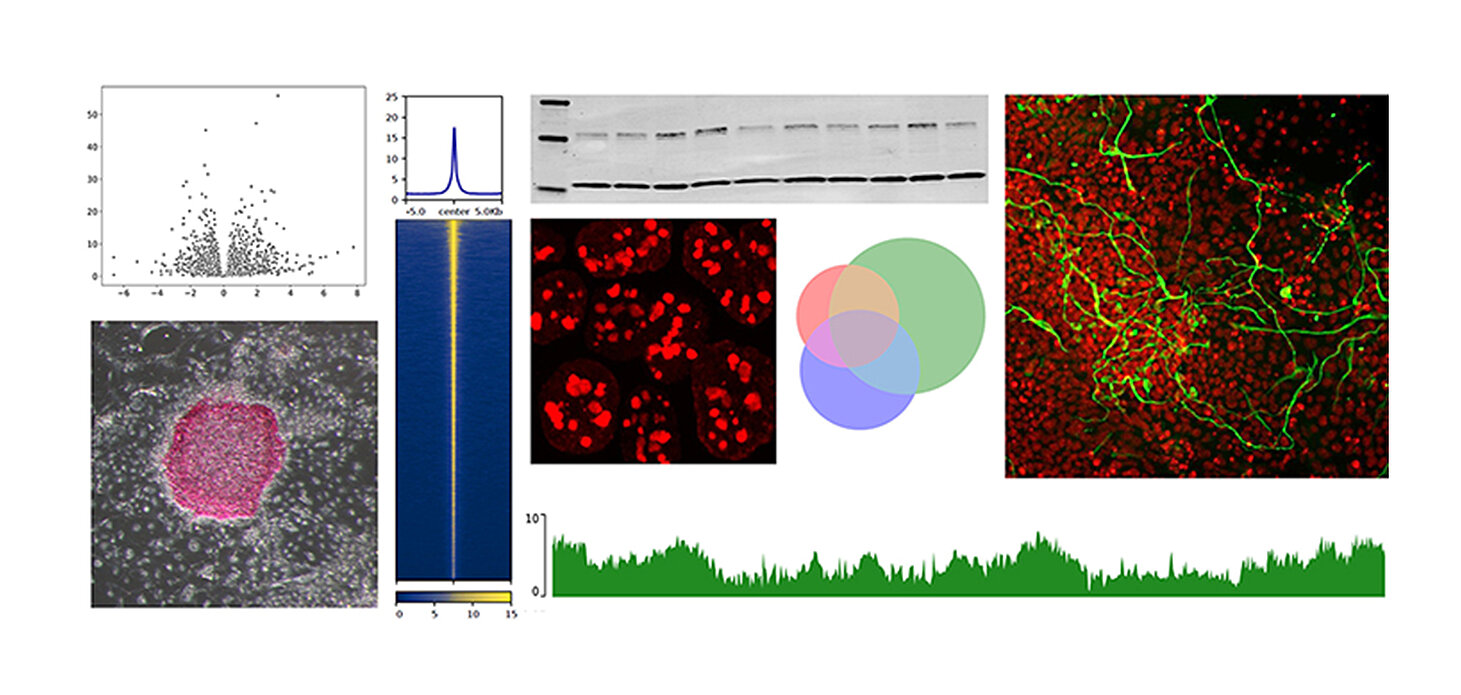
L’ADN est empaqueté sous forme de « chromatine », incluant des nucléosomes, des modifications épigénétiques et de nombreuses protéines associées à divers processus biologiques. Les régulateurs chromatiniens permettent un décodage différent de l'information génétique selon le contexte cellulaire. Ainsi, des programmes d'expression génique uniques sont activés dans chaque type cellulaire, conduisant à des phénotypes distincts et à des fonctions spécialisées.
Comment l’identité cellulaire est-elle régulée durant les processus développementaux, ou perturbée en conditions pathologiques ? Dans notre laboratoire, nous abordons cette question fondamentale grâce à une combinaison de technologies d’ingénierie génétique, de biochimie et de séquençage à haut débit. Nos recherches portent plus particulièrement sur les facteurs de transcription et les régulateurs épigénétiques, qui sont essentiels au développement embryonnaire et souvent dérégulés dans des maladies humaines telles que le cancer et les troubles du (neuro-)développement.
Nous tirons avantage des cellules souches embryonnaires de souris comme modèle-système des décisions de destin cellulaire. L’utilisation de lignées cellulaires génétiquement modifiées nous permet non seulement d’évaluer l’influence de protéines d’intérêt sur le transcriptome et l’épigénome, mais aussi de tester leur impact phénotypique sur la différenciation cellulaire in vitro ou le développement embryonnaire in vivo.

PUBLICATIONS RECENTES
Tetramerisation governs SALL transcription factor function in development and disease. S. Giuliani, K. Chhatbar, M. Wear, J. Guy, T. Mathieson, H. Burdett, L. George, G. Alston, C. Spanos, T. McHugh, D. Kelly, R. Pantier and A. Bird. bioRxiv (preprint), 2025. https://doi.org/10.1101/2025.10.27.684836
TET knockout cells transit between pluripotent states and exhibit precocious germline entry. R. Pantier, E. Barbieri, S. Gonzalez Brito, E. Thomson, T. Tatar, D. Colby, M. Zhang, I. Chambers. The EMBO Journal, 2025. https://doi.org/10.1038/s44318-025-00597-9
MeCP2 binds to methylated DNA independently of phase separation and heterochromatin organisation. R. Pantier, M. Brown, S. Han, K. Paton, S. Meek, T. Montavon, N. Shukeir, T. McHugh, D.A. Kelly, T. Hochepied, C. Libert, T. Jenuwein, T. Burdon, A. Bird. Nature Communications, 2024. https://doi.org/10.1038/s41467-024-47395-1
Structure of SALL4 zinc finger domain reveals link between AT-rich DNA binding and Okihiro syndrome. J.A. Watson, R. Pantier, U. Jayachandran, K. Chhatbar, B. Alexander-Howden, V. Kruusvee, M. Prendecki, A. Bird, A.G. Cook. Life Science Alliance, 2023. https://doi.org/10.26508/lsa.202201588
High-throughput sequencing SELEX for the determination of DNA-binding protein specificities in vitro. R. Pantier, K. Chhatbar, G. Alston, H.Y. Lee, A. Bird. STAR Protocols, 2022. https://doi.org/10.1016/j.xpro.2022.101490
SALL4 controls cell fate in response to DNA base composition. R. Pantier, K. Chhatbar, T. Quante, K. Skourti-Stathaki, J. Cholewa-Waclaw, G. Alston, B. Alexander-Howden, H.Y. Lee, A.G. Cook, C.G. Spruijt, M. Vermeulen, J. Selfridge, A. Bird. Molecular Cell, 2021. https://doi.org/10.1016/j.molcel.2020.11.046
TET1 Interacts Directly with NANOG via Independent Domains Containing Hydrophobic and Aromatic Residues. R. Pantier, N. Mullin, E. Hall-Ponsele, I. Chambers. Journal of Molecular Biology, 2020. https://doi.org/10.1016/j.jmb.2020.10.008
Endogenous epitope-tagging of Tet1, Tet2 and Tet3 identifies TET2 as a naïve pluripotency marker. R. Pantier, T. Tatar, D. Colby, I. Chambers. Life Science Alliance, 2019. https://doi.org/10.26508/lsa.201900516
Membres
Chercheur(euse)s
Doctorant(e)s
Projets en cours
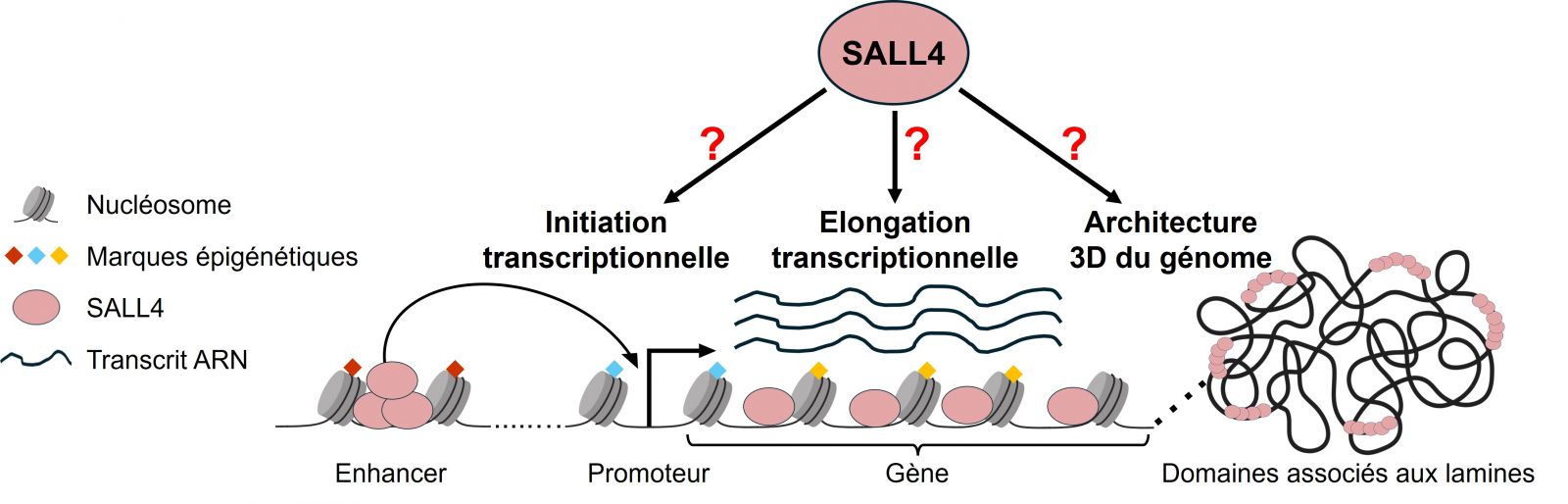
Définition des principes de régulation de l’expression génique par le facteur de transcription atypique SALL4 (AT-BASE)
Nos travaux récents ont démontré que le facteur de pluripotence SALL4 contrôle la transcription proportionnellement à la teneur en nucléotides A/T de ses gènes cibles. Ce projet vise à disséquer les mécanismes moléculaires impliqués dans ce nouveau mode de régulation de l’expression génique à trois échelles génomiques différentes :
- Enhancers : régulation de l'initiation de la transcription via des interactions à longue distance avec les promoteurs de gènes cibles ?
- Corps de gènes : régulation de l'élongation de la transcription via une concentration locale de SALL4 sur les unités géniques ?
- Domaines associés aux lamines : régulation de l'architecture génomique tridimensionnelle via une liaison globale de SALL4 sur les régions riches en nucléotides A/T ?
Financements et partenaires
- 2025: Programme « Jeunes Chercheurs et Jeunes Chercheuses » (JCJC) – Agence Nationale de la Recherche (ANR)
- 2025: Financement « Tremplin-ERC » (T-ERC) – Agence Nationale de la Recherche (ANR)
- 2025: Initiative d’Excellence (IdEx) « Attractivité » – Université de Strasbourg
- 2025: Financement initial Laboratoire d’Excellence (LabEx) – IGBMC




Evénements
Le
13/042026

