Biologie des Systèmes aléatoires de la régulation des gènes
Biologie des Systèmes aléatoires de la régulation des gènes
La régulation des gènes est un processus réalisé en plusieurs étapes jouant un rôle central dans l'élaboration de la réponse cellulaire à des stimuli environnementaux. Premièrement, les facteurs de transcription pionniers (TFs) reconnaissent les sites de liaison des séquences d'ADN spécifiques dans les régions promotrices de gènes et des sites de régulation. Par la suite, des enzymes spécifiques sont recrutés, modifient les queues d'histones post-traductionnel et provoquent le remodelage des nucléosomes. Les TFs supplémentaires se lient dans les régions de chromatine accessibles et enfin, l'ARN polymérase peut lancer la synthèse de l'ARNm. Ces réactions se produisent dans le noyau compact où la chromatine forme une structure complexe en 3D. Et finalement, les fluctuations aléatoires font que la dynamique de l'expression des gènes dans des cellules individuelles diffère considérablement du comportement moyen au niveau de la population.
Les nouvelles technologies expérimentales nous permettent de mesurer, les interactions protéine-ADN, les modifications post-transcriptionnel des histones et de la structure 3D de la chromatine dans le génome entier au sein des populations et plus récemment dans des cellules individuelles. En utilisant ces méthodes d’expériences Time-cours, nous présentons une occasion unique d'étudier, d'une manière quantitative, la dynamique de la régulation des gènes.
La principale activité de recherche de notre groupe est de construire des modèles biophysiques aléatoires de la régulation des gènes eucaryotes en intégrant tous ces ensembles de données. Nous utilisons des méthodes fondées sur des principes ancrés dans la bio-informatique, la statistique bayésienne (démarche logique permettant de calculer ou réviser la probabilité d'une hypothèse.), les processus aléatoires et la mécanique statistique.
Membres
Chercheur(euse)s
Post-doctorant(e)s
Doctorant(e)s
Ingénieur(eure)s
Publications
-
2025
-
Biophysical Modeling Uncovers Transcription Factor and Nucleosome Binding on Single DNA Molecules
- Lasha Dalakishvili
- Husain Managori
- Anaïs Bardet
- Věra Slaninová
- Edouard Bertrand
- Nacho Molina
-
Gene-specific transcript buffering revealed by perturbation of coactivator complexes
- Faezeh Forouzanfar
- David F Moreno
- Damien Plassard
- Audrey Furst
- Karen Amaral de Oliveira
- Bernardo Reina San Martin
- Làszlo Tora
- Nacho Molina
- Manuel Mendoza
Science Advances ; Volume: 11 ; Page: eadr1492
-
Mouse promoters are characterised by low occupancy and high turnover of RNA polymerase II
- Kasit Chatsirisupachai
- Christina J. I. Moene
- Rozemarijn Kleinendorst
- Elisa Kreibich
- Nacho Molina
- Arnaud Krebs
Molecular Systems Biology ; Volume: 21 ; Page: 447 - 471
-
-
2024
-
Gene-specific RNA homeostasis revealed by perturbation of the NuA4/Tip60 acetyltransferase complex
- Faezeh Forouzanfar
- Damien Plassard
- Damien Plassard
- Audrey Furst
- David F Moreno
- Karen A Oliveira
- Bernardo Reina-San-Martin
- László Tora
- Nacho Molina
- Manuel Mendoza
-
Single-cell multiomics reveals the oscillatory dynamics of mRNA metabolism and chromatin accessibility during the cell cycle
- Maulik K Nariya
- David Santiago-Algarra
- Olivier Tassy
- Marie Cerciat
- Tao Ye
- Andrea Riba
- Nacho Molina
-
Transcription processes compete with loop extrusion to homogenize promoter and enhancer dynamics
- Angeliki Platania
- Cathie Erb
- Mariano Barbieri
- Bastien Molcrette
- Erwan Grandgirard
- Marit de Kort
- Wim Pomp
- Karen Meaburn
- Tiegh Taylor
- Virlana Shchuka
- Silvia Kocanova
- Mariia Nazarova
- Guilherme Monteiro Oliveira
- Jennifer Mitchell
- Evi Soutoglou
- Tineke Lenstra
- Nacho Molina
- Argyris Papantonis
- Kerstin Bystricky
- Tom Sexton
Science Advances ; Volume: 10 ; Page: eadq0987
-
-
2023
-
Characterization of cell-fate decision landscapes by estimating transcription factor dynamics
- Sara Jiménez
- Valérie Schreiber
- Reuben Mercier
- Gérard Gradwohl
- Nacho Molina
Cell Reports Methods ; Page: 100512
-
Competition between transcription and loop extrusion modulates promoter and enhancer dynamics
- Angeliki Platania
- Cathie Erb
- Mariano Barbieri
- Bastien Molcrette
- Erwan Grandgirard
- Marit Ac de Kort
- Karen Meaburn
- Tiegh Taylor
- Virlana M Shchuka
- Silvia Kocanova
- Guilherme Monteiro Oliveira
- Jennifer A Mitchell
- Evi Soutoglou
- Tineke L Lenstra
- Nacho Molina
- Argyris Papantonis
- Kerstin Bystricky
- Tom Sexton
- Guilherme Monteiro Oliveira
-
-
2022
-
Cell cycle gene regulation dynamics revealed by RNA velocity and deep-learning
- Andrea Riba
- Attila Oravecz
- Matej Durik
- Sara Jiménez
- Violaine Alunni
- Marie Cerciat
- Matthieu Jung
- Céline Keime
- William M Keyes
- Nacho Molina
Nature Communications ; Volume: 13 ; Page: 2865
-
-
2021
-
Precise measurements of chromatin diffusion dynamics by modeling using Gaussian processes
- Guilherme Oliveira
- Attila Oravecz
- Dominique Kobi
- Manon Maroquenne
- Kerstin Bystricky
- Thomas Sexton
- Nacho Molina
Nature Communications ; Volume: 12 ; Page: 6184
-
Page 1 sur 2
Prix/Distinctions
2021 | USIAS Fellowship. University of Strasbourg Institute for Advanced Study, France.
2021 | Theory@EMBL Fellowship. EMBL Heidelberg, Germany.
2016 | IDEX Chair for Attracting Research. University of Strasbourg, France.
2016 | INRT LabEx Chair of Excellence. IGBMC. France.
2013 | Chancellor’s Fellowship. University of Edinburgh, UK.
2011 | SIB Young Bioinformatician Award. Swiss Institute of Bioinformatics, Switzerland.
Actualités
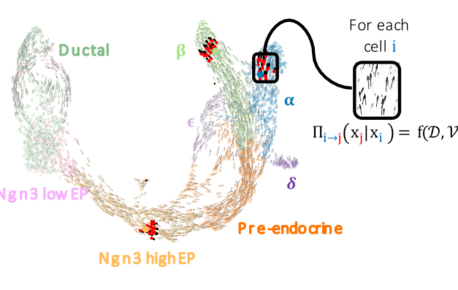
FateCompass : identifier et prédire les critères décisifs du destin cellulaire
La différenciation cellulaire est un processus régulé par l’expression des gènes grâce à l’action de facteurs de transcription. Différents facteurs de…
Lire la suite

