Organisation spatiale du génome
Organisation spatiale du génome
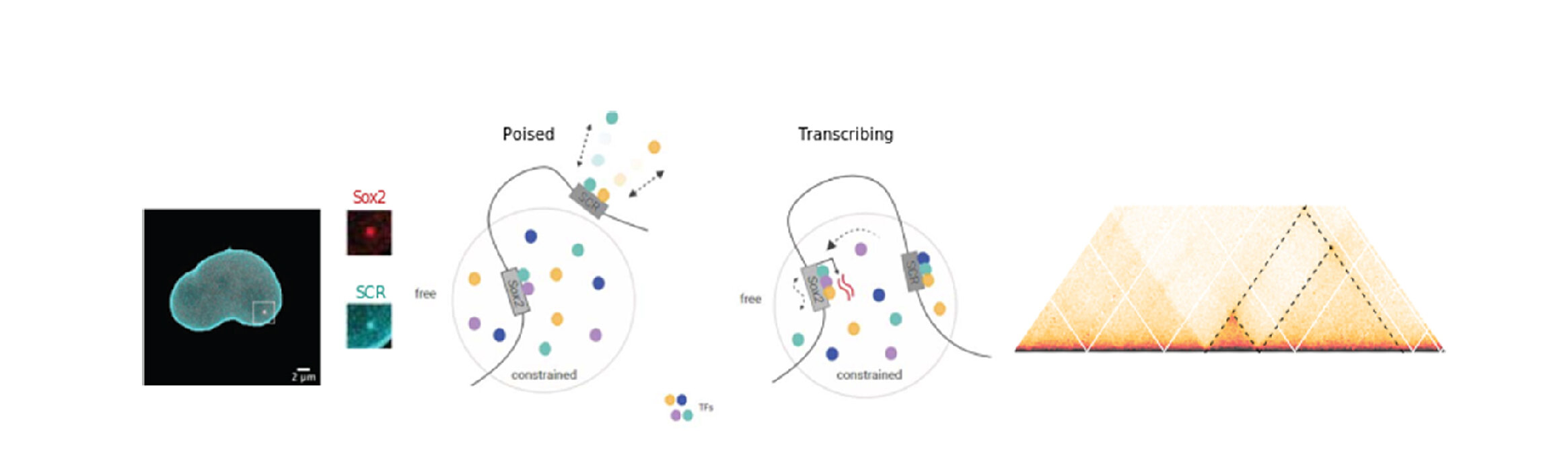
Chaque cellule du corps humain doit compacter deux mètres d'ADN dans un noyau d'environ dix micromètres, ce qui équivaut à faire tenir la Tour Eiffel dans une pièce de un centime. D'une manière ou d'une autre, ceci est réalisé de façon à ce que les gènes nécessaires à chaque cellule soient accessibles à la machinerie pour leur activation. Notre équipe combine des approches génomiques (tels qu Hi-C et ses variantes), l'édition du génome par CRISPR-Cas9 et la microscopie à haute résolution temporelle pour disséquer l'organisation du génome dans l'espace et le temps, et pour voir comment elle est responsable du contrôle de l'expression des gènes.
Twitter : @TomSexton_lab
Membres
Chercheur(euse)s
Doctorant(e)s
Ingénieur(eure)s
Collaborateur(trice)s occasionnel(le)s
Anciens membres
Yousra Ben Zouari (PhD) – en postdoc au FMI, Bâle.
Nezih Karasu (PhD) – en postdoc à l’IGMM, Montpellier
Angeliki Platania (PhD)
Sanjay Chahar (postdoc) – en postdoc a l’IGMM, Montpellier
Anne Molitor (postdoc) – poste d’Ingénieur de Recherche à l’Institut d’Immuno-Rhumathologie Moléculaire, Strasbourg
Natalia Sikorska (Postdoc) – suit une formation médicale
Gui Oliveira (Postdoc) – a accédé à un poste d'ingénieur logiciel chez MathWorks
Dominique Kobi (ingénieur) – a accédé à un poste chez Machery-Nagel
Manon Maroquenne (ingénieur) – a accédé à un poste au laboratoire de Bioingénerie et Bio-imagerie des tissus ostéo-articulaires, Paris
Audrey Mossler (ingénieur) – est au Imperial College, UK
Projets en cours
Nous nous intéressons à tous les aspects de l'organisation 3D ou 4D du génome qui influencent l'expression des gènes, mais nos recherches actuelles peuvent être divisées en deux axes principaux :
- Des approches de biologie moléculaire à l'échelle du génome (par exemple Capture Hi-C) pour caractériser les changements épigénomiques accompagnant les transitions développementales et les réponses aux traitements chimiothérapeutiques.
- L’étiquetage de loci génomiques spécifiques et leur suivi en temps réel pour voir comment la mobilité de la chromatine affecte (et est affectée par) la transcription et les réponses de réparation de l'ADN.
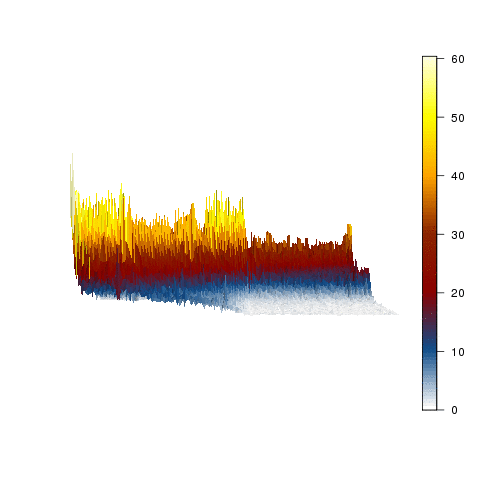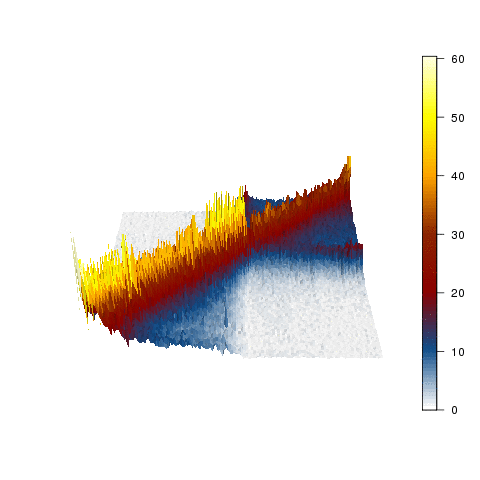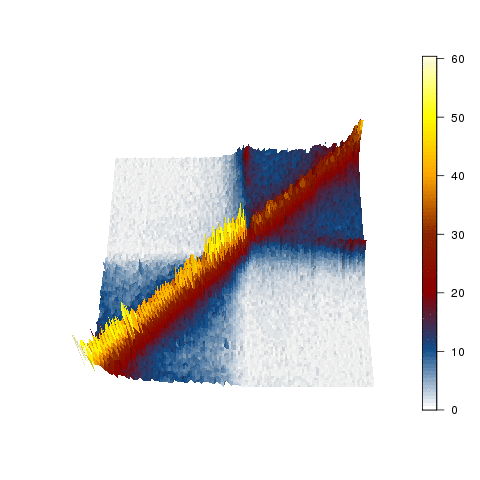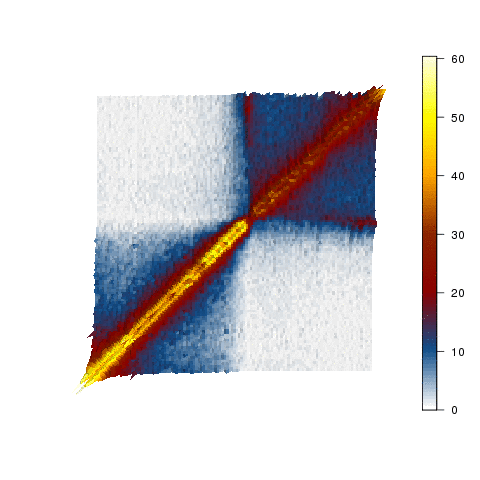
Notre première tentative de modélisation du noyau des cellules souches.
Collaborations et réseaux
A l’IGBMC
- Nacho Molina
- Daniel Metzger
A Strasbourg
- Karine Merienne (Laboratoire de Neurosciences Cognitives et Adaptives)
- Jean-Noel Freund/Claire Domon-Dell (Interface de Recherche Fondamentale et Appliquée Research en Cancerologie)
En France
- Kerstin Bystricky (Centre de Biologie Intégrative, Toulouse)
- Christophe Ginestier/Emmanuelle Charafe-Jauffret (Centre de Cancérologie de Marseille, Marseille)
- Mounia Lagha (Institut de génétique moléculaire, Montpellier)
- Reini Luco (Institut Curie, Paris)
- Salvatore Spicuglia (Théories and Approches de la complexité génomique, Marseille)
- Jean-Christophe Andrau (Institut de génétique moléculaire, Montpellier)
- Daniel Jost (Ecole Normale Superieur, Lyon)
A l’international
- Jennifer Mitchell (University of Toronto, Canada)
- Tineke Lenstra (National Cancer Institute, Netherlands)
- Akis Papantonis (University of Goettingen, Germany)
- Evi Soutoglou (Genome Damage and Stability Centre, UK)
Financements et partenaires
ERC Starting Grant 2015-2021;
ATIP-Avenir 2014-2017;
ANR; INCa; USIAS
Prix/Distinctions
Prix Claude Paoletti, 2015
Ressources
Voir nos outils Github
Pour l’analyse et l’exploration de Capture Hi-C (ChiCMaxima)
Pour l’exploration des données de 4C (4See)
Pour la mesure des propriétés de diffusion à partir des résultats d’imagerie en temps réél (GPTool)
Publications
-
2017
-
Chromosome topology guides the Drosophila Dosage Compensation Complex for target gene activation
- Tamás Schauer
- Yad Ghavi-Helm
- Tom Sexton
- Christian Albig
- Catherine Regnard
- Giacomo Cavalli
- Eileen Em Furlong
- Peter Becker
EMBO Reports ; Volume: 18 ; Page: 1854-1868
-
Detecting Spatial Chromatin Organization by Chromosome Conformation Capture II: Genome-Wide Profiling by Hi-C
- Matteo Viettri Rudan
- Suzana Hadjur
- Thomas Sexton
Methods in Molecular Biology ; Volume: 1589 ; Page: 47-74
-
Page 3 sur 3
