Corégulateurs des récepteurs nucléaires dans la spermatogenèse
Responsable de sous-groupe : Nadege VERNET
Équipe(s) : Physiopathologie des voies de signalisation de la vitamine A
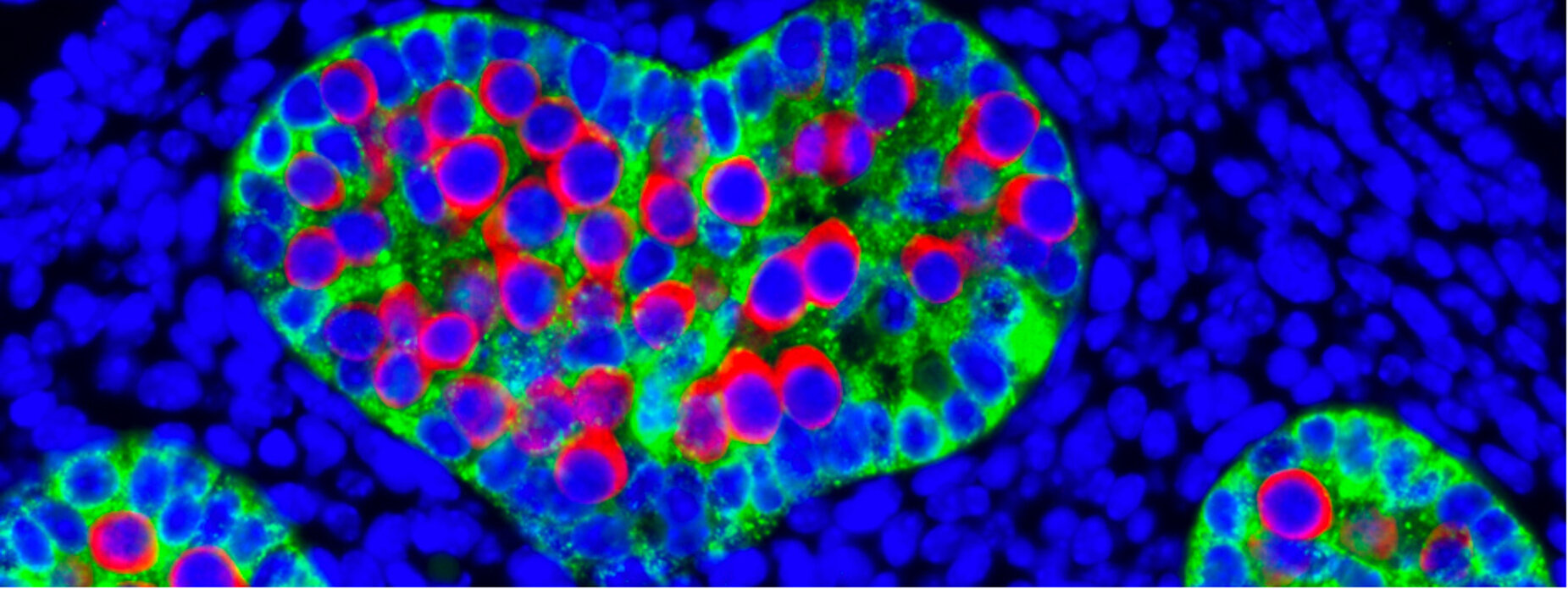
L’acide rétinoïque (ATRA), le métabolite actif de la vitamine A, est produit localement au niveau de ses tissus cibles. Il est indispensable au développement embryonnaire et à la reproduction. Ses dérivés pharmaceutiques sont des outils thérapeutiques au potentiel prometteur. Notre projet consiste à décrypter, étape par étape, les processus dépendants de l’ATRA impliqués dans la différenciation des cellules in vivo, en nous focalisant sur les cellules germinales (CG) de la souris, à l’origine des gamètes, comme modèle d’étude.
La chronologie de la gamétogenèse chez les mammifères varie selon le sexe. Les femelles naissent avec une réserve définitive de CG, dont le nombre conditionne la fertilité. Chez les mâles, la fertilité repose sur un équilibre permanent entre auto-renouvèlement et différenciation des CG. Dans les deux sexes, des précurseurs indifférenciés se divisent pour augmenter la population de CG. Celles-ci entrent ensuite en méiose, réalisant la recombinaison génétique et la réduction de moitié du nombre de chromosomes nécessaire à la production de gamètes haploïdes. Des études génétiques et pharmacologiques chez la souris ont révélé que l’ATRA et ses récepteurs nucléaires (RAR) sont des acteurs majeurs de la gamétogenèse. Notre projet vise à décrypter, étape par étape, les processus dépendants de l’ATRA et requis pour la différenciation des CG in vivo, depuis les cellules souches capables d’auto-renouvèlement jusqu’aux gamètes aptes à la fécondation, en passant par l’étape de méiose grâce à laquelle les chromosomes sont brassés à chaque génération pour assurer la diversité génétique des individus. Pour ce faire, nous disposons d’une collection de souris, unique au monde par sa diversité, chez lesquelles un ou plusieurs des acteurs de la voie de signalisation de l’ATRA sont invalidés, ainsi que de l’expertise scientifique et du savoir-faire pour caractériser les anomalies tissulaires et moléculaires.
A terme, notre projet permettra de mieux comprendre les pathologies gonadiques de l’Homme (par exemple, ménopauses précoces, défauts de production des spermatozoïdes) et le cas échéant de proposer aux patients infertiles des traitements à base des rétinoïdes (avec des agonistes ou antagonistes). Par ailleurs, la possibilité de perturber transitoirement la signalisation de l’ATRA représente une stratégie prometteuse pour développer des contraceptifs masculins, à condition que le mécanisme du fonctionnement de l'ATRA soit totalement déchiffré pour pallier les effets secondaires.
Au-delà de la gamétogenèse, notre projet permettra aussi de comprendre la manière dont l’ATRA contrôle la différenciation de cellules souches, qu’elles soient normales ou cancéreuses.
 | Déconstruire et reconstruire les embryonshttps://moodle.unistra.fr/course/view.php?id=13647 Choisir: connexion anonyme Mot de passe: ontogenesis |
 Valérie DUPE (CRCN Inserm), ORCID ID orcid.org/0000-0003-4859-0612, Institut de Génétique et Développement de Rennes (IGDR, UMR 6290 CNRS) , Rennes, France. Equipe Génétique des pathologies du développement du neurectoderme https://igdr.univ-rennes1.fr/genetique-des-pathologies-du-developpement-du-neurectoderme
Valérie DUPE (CRCN Inserm), ORCID ID orcid.org/0000-0003-4859-0612, Institut de Génétique et Développement de Rennes (IGDR, UMR 6290 CNRS) , Rennes, France. Equipe Génétique des pathologies du développement du neurectoderme https://igdr.univ-rennes1.fr/genetique-des-pathologies-du-developpement-du-neurectoderme
Olivia W ENDLING (IR, CNRS), Institut Clinique de la Souris (ICS), Département de Phénotypage, Illkirch, France ? Equipe Histopathologie et Embryologie http://www.ics-mci.fr/en/departments/phenotyping/histopathology-and-embryology/
ENDLING (IR, CNRS), Institut Clinique de la Souris (ICS), Département de Phénotypage, Illkirch, France ? Equipe Histopathologie et Embryologie http://www.ics-mci.fr/en/departments/phenotyping/histopathology-and-embryology/
 Nicolas MATT (PU UNISTRA), Institut de Biologie Moléculaire et Cellulaire de Strasbourg (IBMC-M3i, UPR 9022 CNRS), Strasbourg, France. Equipe Génétique de la réponse immunitaire ibmc.cnrs.fr/en/laboratoire/m3i-en/equipes/genetics-of-immune-response/, Vice-Président Patrimoine UNISTRA www.unistra.fr/equipe-presidentielle/nicolas-matt
Nicolas MATT (PU UNISTRA), Institut de Biologie Moléculaire et Cellulaire de Strasbourg (IBMC-M3i, UPR 9022 CNRS), Strasbourg, France. Equipe Génétique de la réponse immunitaire ibmc.cnrs.fr/en/laboratoire/m3i-en/equipes/genetics-of-immune-response/, Vice-Président Patrimoine UNISTRA www.unistra.fr/equipe-presidentielle/nicolas-matt
 Bénédicte MASCREZ (Associate scientist), Université de Genève, Département de Génétique et Evolution, Geneva, Swiss. Equipe Génétique du Développement genev.unige.ch/research/people/Benedicte-Mascrez
Bénédicte MASCREZ (Associate scientist), Université de Genève, Département de Génétique et Evolution, Geneva, Swiss. Equipe Génétique du Développement genev.unige.ch/research/people/Benedicte-Mascrez
 Frank SCHNUTGEN (Lab manager), ORCID ID orcid.org/0000-0002-2426-6685, Klinikum der Johann Wolfgang Goethe-Universität Frankfurt, Frankfurt am Main, Hessen, Germany. Serve Lab www.uniklinik-onkologie.de/fuer-scientists/research-groups/serve-lab/contact/
Frank SCHNUTGEN (Lab manager), ORCID ID orcid.org/0000-0002-2426-6685, Klinikum der Johann Wolfgang Goethe-Universität Frankfurt, Frankfurt am Main, Hessen, Germany. Serve Lab www.uniklinik-onkologie.de/fuer-scientists/research-groups/serve-lab/contact/
 Hugues JACOB (IR, CERBM-GIE), Institut Clinique de la Souris (ICS), Département de Phénotypage, Illkirch, France. Equipe Histopathologie et Embryologie www.ics-mci.fr/en/departments/phenotyping/histopathology-and-embryology/
Hugues JACOB (IR, CERBM-GIE), Institut Clinique de la Souris (ICS), Département de Phénotypage, Illkirch, France. Equipe Histopathologie et Embryologie www.ics-mci.fr/en/departments/phenotyping/histopathology-and-embryology/
 Mathilde RAVERDEAU (post-doctorante), ORCID ID orcid.org/0000-0002-6088-6902, Trinity College Dublin, School of Biochemistry and Immunology, , Dublin, Ireland. Team Immune regulation research www.tcd.ie/Biochemistry/research/mills/
Mathilde RAVERDEAU (post-doctorante), ORCID ID orcid.org/0000-0002-6088-6902, Trinity College Dublin, School of Biochemistry and Immunology, , Dublin, Ireland. Team Immune regulation research www.tcd.ie/Biochemistry/research/mills/
 Aurore GELY-PERNOT (MCU EHESP), ORCID ID: orcid.org/0000-0002-7483-3297, Institut de recherche en santé, environnement et travail (IRSET, Inserm UMR 1085), Rennes, France. Equipe Physiologie et physiopathologie du tractus uro-génital www.irset.org/interlocuteurs/gely-pernot-aurore et Ecole des hautes études en santé publique (EHESP), Département santé-environnement-travail et génie sanitaire (DSETGS), Rennes, France https://www.ehesp.fr/annuaire/enseignement-recherche/aurore-gely-pernot/
Aurore GELY-PERNOT (MCU EHESP), ORCID ID: orcid.org/0000-0002-7483-3297, Institut de recherche en santé, environnement et travail (IRSET, Inserm UMR 1085), Rennes, France. Equipe Physiologie et physiopathologie du tractus uro-génital www.irset.org/interlocuteurs/gely-pernot-aurore et Ecole des hautes études en santé publique (EHESP), Département santé-environnement-travail et génie sanitaire (DSETGS), Rennes, France https://www.ehesp.fr/annuaire/enseignement-recherche/aurore-gely-pernot/
 Sirine SOUALI-CRESPO (post-doctorante)
Sirine SOUALI-CRESPO (post-doctorante)
Responsable de sous-groupe : Nadege VERNET
Équipe(s) : Physiopathologie des voies de signalisation de la vitamine A
 Gérard BENOIT, ORCID ID orcid.org/0000-0003-3148-4127 Laboratoire de Biologie et Modélisation de la Cellule (LBMC, CNRS UMR 5239), Ecole Normale Supérieur, Lyon, France. Equipe Systems Biology of Decision Making, Axe 2 LITREA: Deciphering nuclear receptor biological function in differentiating mouse pluripotent embryonic cells www.ens-lyon.fr/LBMC/equipes/systems-biology-of-decision-making/themes-de-recherche/deciphering-nuclear-receptor-biological-function-in-differentiating-mouse-pluripotent-embryonic-cells
Gérard BENOIT, ORCID ID orcid.org/0000-0003-3148-4127 Laboratoire de Biologie et Modélisation de la Cellule (LBMC, CNRS UMR 5239), Ecole Normale Supérieur, Lyon, France. Equipe Systems Biology of Decision Making, Axe 2 LITREA: Deciphering nuclear receptor biological function in differentiating mouse pluripotent embryonic cells www.ens-lyon.fr/LBMC/equipes/systems-biology-of-decision-making/themes-de-recherche/deciphering-nuclear-receptor-biological-function-in-differentiating-mouse-pluripotent-embryonic-cells
 Marie-Christine CHABOISSIER, ORCID ID orcid.org/0000-0003-0934-8217 Institut de Biologie Valrose (iBV, INSERM U1091, CNRS UMR 7277), Université Nice-Côte d’Azur, Nice, France. Equipe Détermination du sexe chez la souris ibv.unice.fr/research-team/chaboissier/
Marie-Christine CHABOISSIER, ORCID ID orcid.org/0000-0003-0934-8217 Institut de Biologie Valrose (iBV, INSERM U1091, CNRS UMR 7277), Université Nice-Côte d’Azur, Nice, France. Equipe Détermination du sexe chez la souris ibv.unice.fr/research-team/chaboissier/
 Frédéric CHALMEL, ORCID ID orcid.org/0000-0002-0535-3628 Institut de recherche en santé, environnement et travail (IRSET, INSERM UMR 1085), Rennes, France. Equipe Physiologie et physiopathologie du tractus uro-génital, Responsable Axe 4 : Différenciation gonadique et physiologie des gonades www.irset.org/axe-4-differentiation-gonadique-et-physiologie-des-gonades
Frédéric CHALMEL, ORCID ID orcid.org/0000-0002-0535-3628 Institut de recherche en santé, environnement et travail (IRSET, INSERM UMR 1085), Rennes, France. Equipe Physiologie et physiopathologie du tractus uro-génital, Responsable Axe 4 : Différenciation gonadique et physiologie des gonades www.irset.org/axe-4-differentiation-gonadique-et-physiologie-des-gonades
 Gregg DUESTER, ORCID ID orcid.org/0000-0003-4335-3650 Center for Genetic Disorders and Aging Research, Sanford Burnham Prebys Medical Discovery Institute (SBP), Development, Aging and Regeneration Program, La Jolla, California, USA. Team leader www.sbpdiscovery.org/our-scientists/gregg-duester-phd
Gregg DUESTER, ORCID ID orcid.org/0000-0003-4335-3650 Center for Genetic Disorders and Aging Research, Sanford Burnham Prebys Medical Discovery Institute (SBP), Development, Aging and Regeneration Program, La Jolla, California, USA. Team leader www.sbpdiscovery.org/our-scientists/gregg-duester-phd
 Pierre GERMAIN, ORCID ID orcid.org/0000-0003-2962-9593 Centre de Biologie Structurale (CBS, CNRS UMR 5048, INSERM U 1054), Montpellier, France. Equipe Récepteurs nucléaires, intégrateurs de signaux endogènes et environnementaux http://www.cbs.cnrs.fr/index.php/fr/home-equipea5
Pierre GERMAIN, ORCID ID orcid.org/0000-0003-2962-9593 Centre de Biologie Structurale (CBS, CNRS UMR 5048, INSERM U 1054), Montpellier, France. Equipe Récepteurs nucléaires, intégrateurs de signaux endogènes et environnementaux http://www.cbs.cnrs.fr/index.php/fr/home-equipea5
 Gabriel LIVERA, ORCID ID orcid.org/0000-0001-8436-4730 Institut de Radiobiologie Cellulaire et Moléculaire (IRCM CEA), Stabilité Génétique, Cellules Souches et Radiations (UMR SGCSR), Fontenay aux Roses, France. Equipe Laboratoire de Développement des Gonades. jacob.cea.fr/drf/ifrancoisjacob/Pages/Departements/IRCM/Equipes/LDG.aspx
Gabriel LIVERA, ORCID ID orcid.org/0000-0001-8436-4730 Institut de Radiobiologie Cellulaire et Moléculaire (IRCM CEA), Stabilité Génétique, Cellules Souches et Radiations (UMR SGCSR), Fontenay aux Roses, France. Equipe Laboratoire de Développement des Gonades. jacob.cea.fr/drf/ifrancoisjacob/Pages/Departements/IRCM/Equipes/LDG.aspx
 Serge NEF, ORCID ID orcid.org/0000-0001-5462-0676 Université de Genève, Département de Médecine génétique et développement, Genève, Suisse. Equipe Mécanismes moléculaires et génétiques du développement sexuel www.unige.ch/medecine/nef/
Serge NEF, ORCID ID orcid.org/0000-0001-5462-0676 Université de Genève, Département de Médecine génétique et développement, Genève, Suisse. Equipe Mécanismes moléculaires et génétiques du développement sexuel www.unige.ch/medecine/nef/
 Eric PAILHOUX, ORCID ID orcid.org/0000-0001-8012-9143 Biologie de la Reproduction, Environnement, Epigénétique, et Développement (BREED, INRAE UMR 1198), Jouy-en-Josas, France. Equipe Différenciation gonadique et ses perturbations www6.jouy.inrae.fr/breed/Les-Recherches/Les-equipes-de-recherche/Differenciation-gonadique-et-ses-perturbations-DGP
Eric PAILHOUX, ORCID ID orcid.org/0000-0001-8012-9143 Biologie de la Reproduction, Environnement, Epigénétique, et Développement (BREED, INRAE UMR 1198), Jouy-en-Josas, France. Equipe Différenciation gonadique et ses perturbations www6.jouy.inrae.fr/breed/Les-Recherches/Les-equipes-de-recherche/Differenciation-gonadique-et-ses-perturbations-DGP
 Bertrand SERAPHIN, ORCID ID orcid.org/0000-0002-5168-1921 Institut de Génétique et Biologie Moléculaire et Cellulaire (IGBMC, CNRS UMR7104, INSERM U1258), Département de Génomique Fonctionnelle et Cancer, Illkirch, France. Equipe Réseaux et complexes protéiques régulant la dégradation des ARN messagers eucaryotes www.igbmc.fr/rseraphin
Bertrand SERAPHIN, ORCID ID orcid.org/0000-0002-5168-1921 Institut de Génétique et Biologie Moléculaire et Cellulaire (IGBMC, CNRS UMR7104, INSERM U1258), Département de Génomique Fonctionnelle et Cancer, Illkirch, France. Equipe Réseaux et complexes protéiques régulant la dégradation des ARN messagers eucaryotes www.igbmc.fr/rseraphin
 Michaël WEBER, ORCID ID orcid.org/0000-0003-3484-5821 Biotechnologie et Signalisation Cellulaire (BSC, CNRS UMR 7242), ESBS, Illkirch, France. Equipe Régulation épigénétique de l'identité cellulaire bsc.unistra.fr/equipes-de-recherche/equipe-weber/presentation/
Michaël WEBER, ORCID ID orcid.org/0000-0003-3484-5821 Biotechnologie et Signalisation Cellulaire (BSC, CNRS UMR 7242), ESBS, Illkirch, France. Equipe Régulation épigénétique de l'identité cellulaire bsc.unistra.fr/equipes-de-recherche/equipe-weber/presentation/
 Nadège VERNET, médaille de bronze du CNRS en 2021 https://www.cnrs.fr/fr/personne/medailles-de-bronze-2021
Nadège VERNET, médaille de bronze du CNRS en 2021 https://www.cnrs.fr/fr/personne/medailles-de-bronze-2021
Cell Death and Differentiation
Journal of Cell Science ; Volume: 122 ; Page: 2699-709
Developmental Biology ; Volume: 328 ; Page: 315-27
Proceedings of the National Academy of Sciences of the United States of America ; Volume: 106 ; Page: 4272-7
Nucl Recept Signal ; Volume: 7 ; Page: e002
Journal of Reproduction and Fertility ; Volume: 136 ; Page: 619-26
Immunity ; Volume: 29 ; Page: 758-70
Journal of Cell Science ; Volume: 121 ; Page: 3233-42
Developmental Biology ; Volume: 320 ; Page: 140-8
Proceedings of the National Academy of Sciences of the United States of America ; Volume: 105 ; Page: 6765-70
Page 6 sur 12