Variabilité et fonctionalité de la machinerie d’initiation de la transcription au cours de la différenciation et du développement
Responsable de sous-groupe : Stephane VINCENT
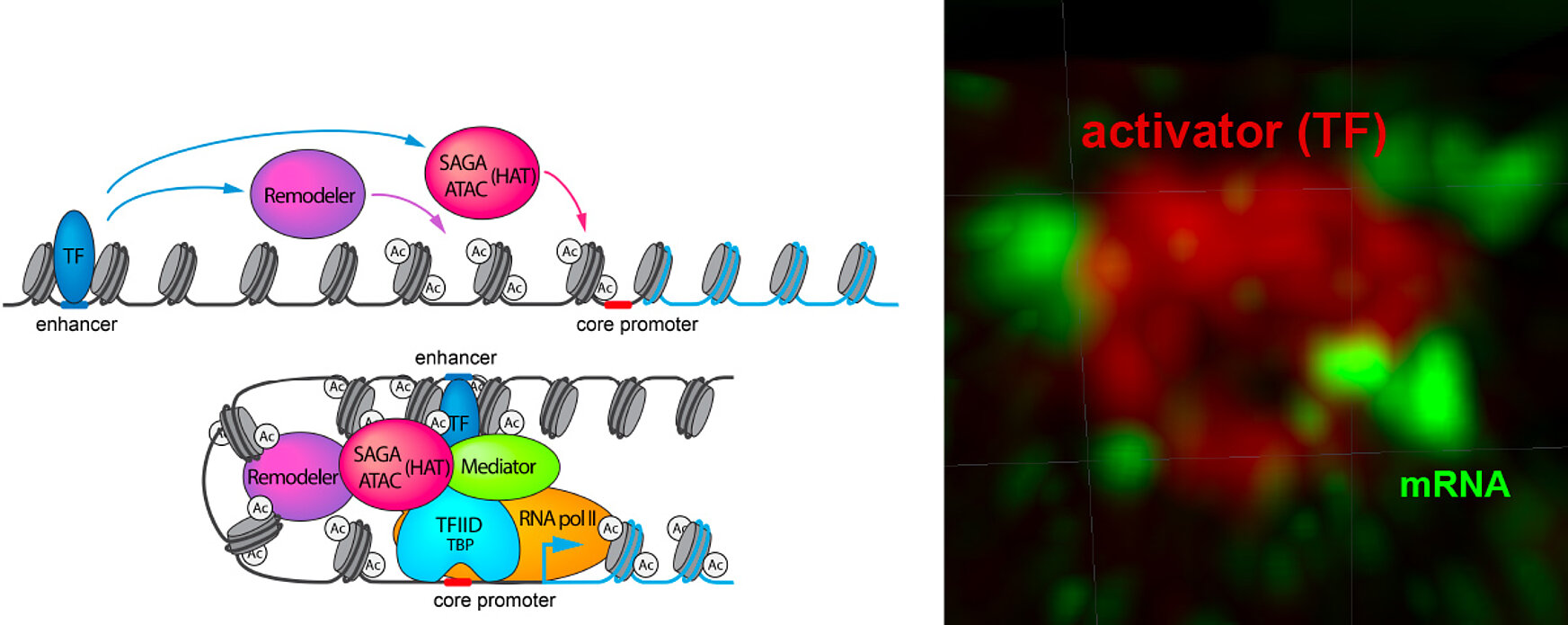
Au cours des dernières années, notre laboratoire a identifié et caractérisé différents facteurs de transcription chez les métazoaires. Ces protéines régulatrices comprennent des facteurs d'initiation de la transcription par l'ARN polymérase II (TFIID en particulier) et des co-activateurs (Spt-Ada-Gcn5 acétyltransférase: SAGA; ou Ada Two A containing, ATAC) qui forment de grands complexes multi-protéiques. Ces complexes sont recrutés aux promoteurs des gènes pour initier la transcription et/ou pour modifier le paysage épigénétique du génome. Nos études démontrent que ces complexes ou leurs variantes jouent un rôle essentiel dans la transcription, ainsi que dans les processus de régulation de la chromatine. Notre objectif principal est de comprendre la nature et les modalités d'assemblage de ces différents complexes, les mécanismes par lesquels ils contrôlent l’expression des gènes codant des protéines dans le noyau d'une cellule donnée, au cours de la croissance, de la différenciation et du développement. Nous utilisons des approches biochimiques, génétiques, de biologie cellulaire, d’imagerie, de biophysique, de bio-informatique et structurales pour étudier le contrôle de l'expression des gènes, la stabilité du génome et l'architecture de la chromatine dans différents organismes eucaryotes (levure, souris et humain). Nos activités visent à mieux comprendre les processus de régulation des gènes dans différents systèmes cellulaires et au cours du développement d'organismes vertébrés. Nous nous intéressons également à la manière dont la perturbation de ces processus hautement régulés est la cause de pathologies.


1) Étudier les mécanismes d'assemblage de différents complexes transcriptionnels;
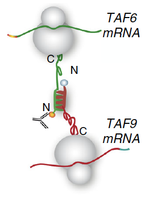
2) Analyser la variabilité de la composition des complexes de pré-initiation et comprendre leur rôle dans la régulation de la transcription pendant le développement et la différenciation (lors de la croissance ovocytaire en particulier);
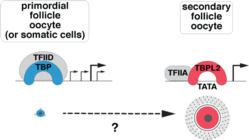
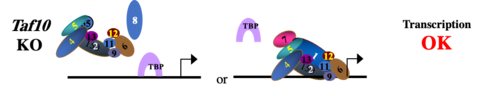
3) Comprendre le rôle des complexes co-activateurs dans la régulation de la transcription;
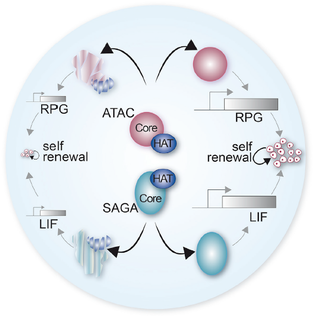
4) Visualiser la transcription par l'ARN polymérase II dans les cellules vivantes au niveau des gènes transcrits;
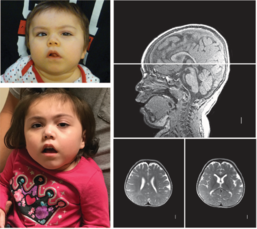
5) Caractériser comment et pourquoi les mutations de certaines sous-unités de ces complexes (TAFs) sont associées à des maladies du système nerveux et des déficiences intellectuelles ?
Research Support for the Tora team in the past 10 years





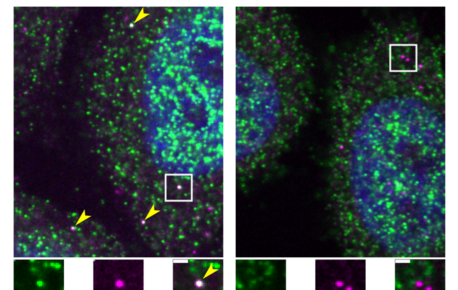
L’activité de nos cellules est régulée par de multiples complexes multiprotéiques, essentiels pour leur fonctionnement. Dans une étude publiée dans…
Lire la suite
Laszlo Tora -







Science Advances ; Volume: 10
Brain - A Journal of Neurology ; Page: Online ahead of print
Journal of Molecular Biology ; Page: 168382
Cell Reports ; Volume: 42
Nature Structural and Molecular Biology ; Volume: 30 ; Page: 1141-1152
Page 1 sur 17